RNA를 isolation하고, RT-PCR을 돌리기 위하여 2일에 걸쳐 실험을 진행하였습니다. RNA isolation은 총 5가지의 단계를 거치는데 homogenization, phase separation, RNA precipitation, RNA washing, Redissociation입니다. Homogenization은 TRIZOL을 이용하여 cell을 파괴하고 RNA를 cell 밖으로 나오게 만듭니다. 바로 isolationmf 하지 않을 경우에는 –70‘C에 보관해야 합니다. phase separation 단계에서는 chloroform을 이용하여 층을 분리시킵니다. incubation 후 centrifuge를 돌릴 때 4’C 온도를 지켜주어야 RNA의 온도에 의한 변성을 막을 수 있습니다. RNA precipitation에서는 DNA, RNA, protein의 분리된 층에서 가운데 투명 부분인 RNA 부분을 조심히 따야 합니다. RNase의 기능이 억제되어 있는 DEPC-etube를 이용하여 실험합니다. 이후 isopropanol을 넣습니다. RNA가 있을 경우에는 흰색의 띠가 나타납니다. 이후 75% EtOH, DEPC-dH2O를 이용하여 RNA washing 후 inverting 합니다. 상등액을 버릴 때에는 아예 뒤집으면 pellet이 딸려나가므로 옆으로 버리고 휴지에 천천히 뒤집습니다. 마지막으로 Redissociation 단계에서 다시 DEPC를 이용하여 pipetting 해준 후 뒤집어서 말립니다. 이 때 뒤집어서 말리는 이유는 물이 날라가지 않고, 에탄올만 날아가게 하기 위해서입니다.
우리가 isolation 한 RNA는 nanodrop을 측정하였을 때 양이 많아 RT-PCR에 사용하지 못하고 새로 sample을 준비하였습니다. LPS 조건에서는 RNA sample을 1.8람다, oligo dT를 1람다, DEPC-dH2O를 7.2람다 넣었으며, LPS+TGF-B 조건에서는 각각 3.2람다, 1람다, 5.8람다씩 넣고 혼합하였습니다. RT-PCR을 돌리기 위해서는 여러 재료들이 필요한데, DEPC-dH2O는 RNase의 activity를 막고, 5XRT-PCR를 완충 용액으로 사용하였습니다. 1람다 X 조건 만큼의 dNTP도 넣고, RNase inhibitor와 Reverse transcriptase도 일정량 넣어주어야 합니다. 반응 후에 꺼내어 ice 조건에서 식혀준 후 각 tube에 mixture를 넣은 후, 37‘C, 1hr, 95’C, 10min에서 반응 시키면 cDNA 합성이 완료됩니다. 여기까지 실험을 진행하면 cDNA가 만들어진 상태이므로 이후에 DEPC tip과 tube를 사용하지 않아도 됩니다. RT-PCR 이후 control을 제공해 주기 위해여 B-actin PCR을 돌립니다.
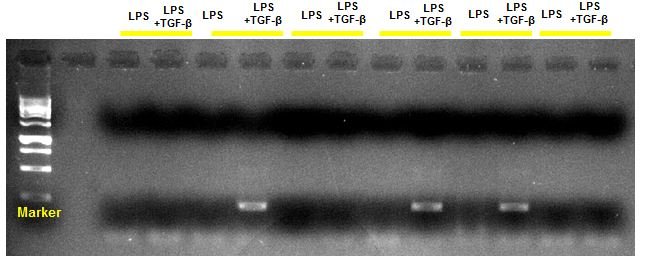
우리는 이 실험을 통해 LPS와 LPS+TGF-B 조건에서 GL-A와 B-actin의 발현량을 비교하기 위하여 PCR 후 band를 내렸습니다. 이 실험에 TGF-B를 이용하는 이유는 class switching 과 관련이 있습니다. 클래스 전환은 항원에 의해 자극도니 성숙한 B세포에서 발생하며, 재배열된 VDJ 유전자가 서로 다른 C 부위의 유전자와 이웃에 위치하게 되는 추가적인 DNA 재배열에 의해 이루어집니다. 클래스 전환은 항원의 자극과 T세포에서 분비되는 사이토카인에 의존합니다. TGF-B가 IgA 항체 합성에 영향을 미치는 것을 조사하는 방법은 총 3가지가 있습니다. 전사수준에서 CA Promoter reporter 조사와 RNA 수준에서 GLA의 발현양을 PCR로 측정하고 단백질 수준에서는 B림프구 표현의 IgA 항체량을 조사하면 됩니다.
